RP2a: In silico Modellierung Cholestase-assoziierter Varianten
Teilprojekt RP2a zielt darauf ab, die molekularen und zellbiologischen Folgen neuartiger Missense-Mutationen, die mittels NGS in RP1 in Zusammenarbeit mit RP2b und RP3 identifiziert wurden, vorherzusagen und zu validieren und sie im Hinblick auf die Auswirkungen der Mutation in Subtypen zu klassifizieren. Der Schwerpunkt liegt dabei auf den Auswirkungen von Missense-Mutationen, da Aminosäure-Substitutionen die größte Variabilität hinsichtlich der Auswirkungen auf Proteinebene aufweisen.
Um eine breitere Nutzung zu gewährleisten und auch für zukünftige Varianten eine Aussage treffen zu können, werden außerdem maschinelle Lernprogramme für spezifische Proteine entwickelt. Aufbauend auf dem Vasor-Projekt aus der ersten HiChol-Förderperiode, mit dem Aminosäure-Substitutionen in dem Transporter MDR3 in benigne oder pathogene Varianten klassifiziert werden können, wird ein Vorhersageprogramm für BSEP-Varianten angestrebt. BSEP transportiert Gallensäuren aus den Leberzellen in die Gallengänge und nimmt dadurch eine zentrale Rolle in der Gallenhomöostase ein. Es wird eigens ein Datensatz für BSEP etabliert basierend auf detaillierten Untersuchungen von Varianten in der wissenschaftlichen Literatur. Anhand dieses Datensatzes wird dann ein maschinelles Lernen-Programm trainiert mit Informationen von neuen sequenz- und strukturbasierten Tools und Prädiktoren, um bestmögliche Vorhersagen für BSEP zu ermöglichen.
Auch für den nuklearen Rezeptor FXR wird ein maschinelles Lernen-basiertes Programm angestrebt. Da für dieses Protein wenige Varianten in der Literatur bekannt sind, wird hier in enger Kollaboration mit RP2b ein Alanin-Scanning durchgeführt für eine Domäne des Proteins, die kritisch für die Protein-Aktivität ist. Hierzu wird jede einzelne Position der Domäne zu der Aminosäure Alanin ausgetauscht und der Effekt auf die Aktivität des Proteins in etablierten zellulären Assays gemessen. Einzelne Varianten, die unerwartete Effekte zeigen oder im klinischen Umfeld identifiziert worden sind, werden durch Molekulardynamik-Simulationen im Detail analysiert auf ihren Einfluss auf konformationsspezifische Interaktionen und die Dynamik des Proteins.
Die durchgeführten Projekte sind eng verknüpft mit den anderen HiChol-Projekten und sind, neben der direkten Bereitstellung von Informationen zu Proteinvarianten, auf die Erstellung von auch zukünftig nutzbaren Tools ausgelegt.
Das Teilprojekt wird an der Heinrich-Heine-Universität Düsseldorf (Projektleitung: Prof. Dr. Holger Gohlke, wissenschaftliche Mitarbeiterin: Dr. Annika Behrendt) bearbeitet.
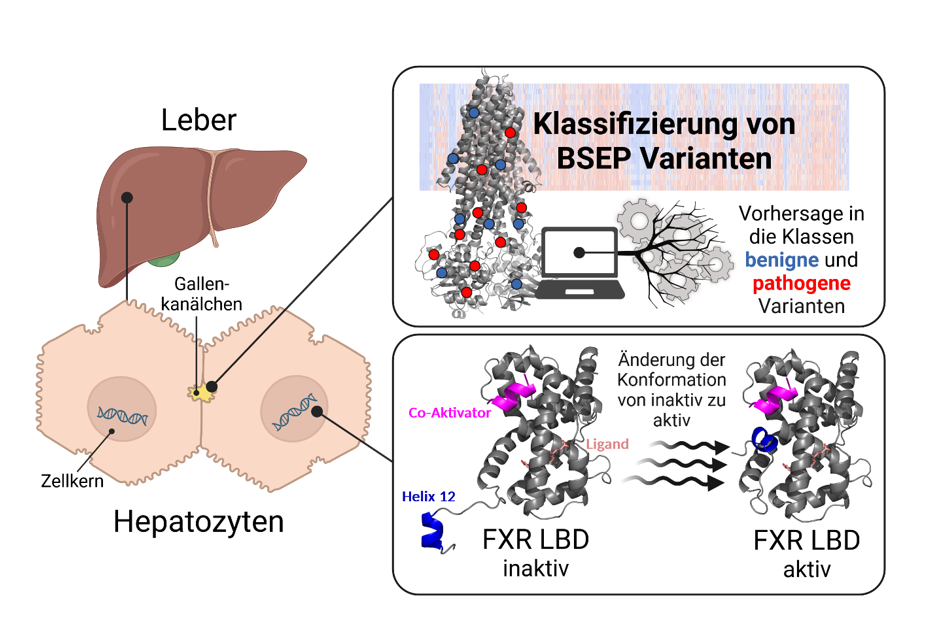
Abbildung 1: In dem Projekt wird ein Vorhersageprogramm für den Gallensäure-Transporter BSEP entwickelt (oben rechts), wobei maschinelles Lernen eingesetzt wird, um auf einem Protein-spezifischen Datensatz zu trainieren und gute Resultate zu ermöglichen. Basierend auf bisherigen neuen Erkenntnissen aus dem HiChol Konsortium, wird die Ligandenbindedomäne (LBD) des nuklearen Rezeptors FXR weiter unter die Lupe genommen (unten rechts), um den Einfluss einzelner Positionen auf die konformationelle Änderung von inaktiv zu aktiv zu bestimmen.
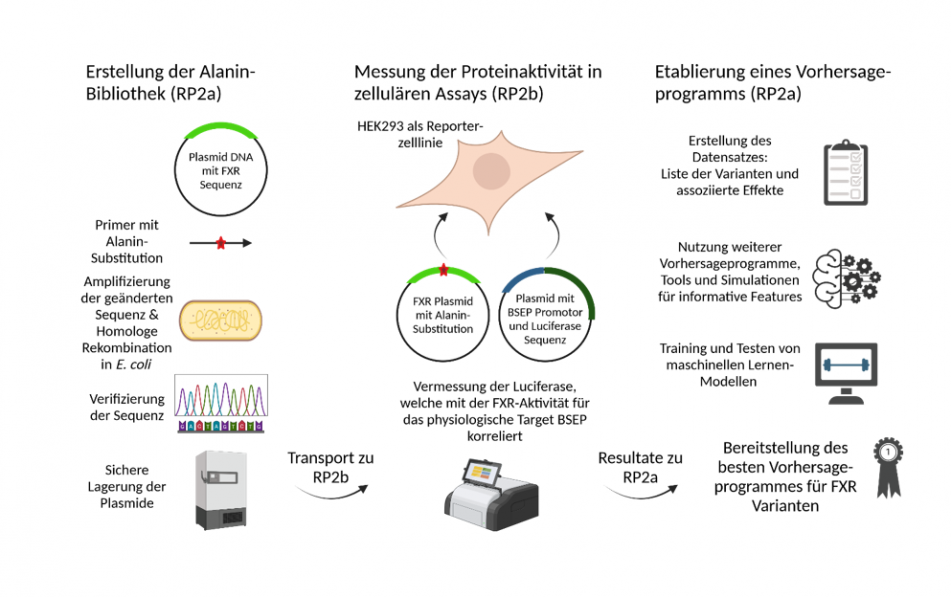
Abbildung 2: Analyse der einzelnen Positionen in FXR, um deren Einfluss auf die Proteinaktivität zu bestimmen und den entstehenden Datensatz für ein maschinelles Lernen-basiertes Programm zu nutzen.
-Abbildungen erstellt unter Nutzung von BioRender-





